Das unerforschte Gebiet der Epitranskriptomik: mRNA-Modifikationen bei psychiatrischen Erkrankungen
Forschungsbericht (importiert) 2018 - Max Planck Institut für Psychiatrie
Stress kann durch epigenetische Mechanismen, wie DNA-Methylierung, die Aktivität von Genen beeinflussen. Neue Forschungsergebnisse zeigen, dass eine ähnliche Regulation auf der Ebene der Botenmoleküle (mRNA) existiert, die für die Übertragung der Erbinformation zuständig sind. Die häufigste mRNA-Modifikation N6-Methyladenosine (m6A) wird durch Stress im Gehirn reguliert und hat eine veränderte Signatur im Blut von Patienten mit Depressionen. Die Erforschung der zugrundeliegenden Mechanismen kann dazu beitragen, die Entstehung stressbedingter psychiatrischer Erkrankungen besser zu verstehen.
Messenger-RNAs (mRNAs) sind Kopien definierter Bereiche unserer DNA, die als Botenmoleküle Erbinformation transportieren: sie übertragen die Information für die Produktion von Proteinen von der DNA an den Ort der Proteinbiosynthese. Abhängig von der Art und dem Bedarf einer Zelle werden unterschiedliche Gene der DNA kopiert, um Proteine und andere funktionale Produkte herzustellen, die es der Zelle ermöglichen, sich an Veränderungen ihrer Umwelt anzupassen. Dieser als Genexpression bezeichnete Prozess wird auf verschiedenen Ebenen reguliert. Auf der Ebene der DNA wird dies durch Modifizierungen der Bausteine oder Struktur der DNA erreicht und führt zu mehr oder weniger RNA-Kopien. Die Mechanismen, die diese Art der Regulation ermöglichen, werden im Forschungsbereich Epigenetik untersucht.
Das neue Forschungsfeld der Epitranskriptomik untersucht Modifikationen der RNA
Die experimentell gemessene Menge an Proteinen ist oft nicht proportional zur Menge vorhandener mRNA [1]. Dies deutet darauf hin, dass es weitere Regulationsmechanismen gibt, die bestimmen, ob und wie effektiv ein mRNA-Molekül zur Proteinproduktion genutzt wird. Ein Mechanismus, der die Feineinstellung der Proteinproduktion auf der Ebene der mRNA ermöglicht, ist die chemische Modifikation der einzelnen mRNA-Bausteine. Der junge Forschungsbereich, der die Funktionen dieser RNA-Modifikationen untersucht, wird Epitranskriptomik genannt. Die in der Epitranskriptomik analysierten RNA-Modifikationen ermöglichen die posttranskriptionelle Regulation der Genexpression: die Beeinflussung der Genaktivität durch Feineinstellung des RNA-Metabolismus.

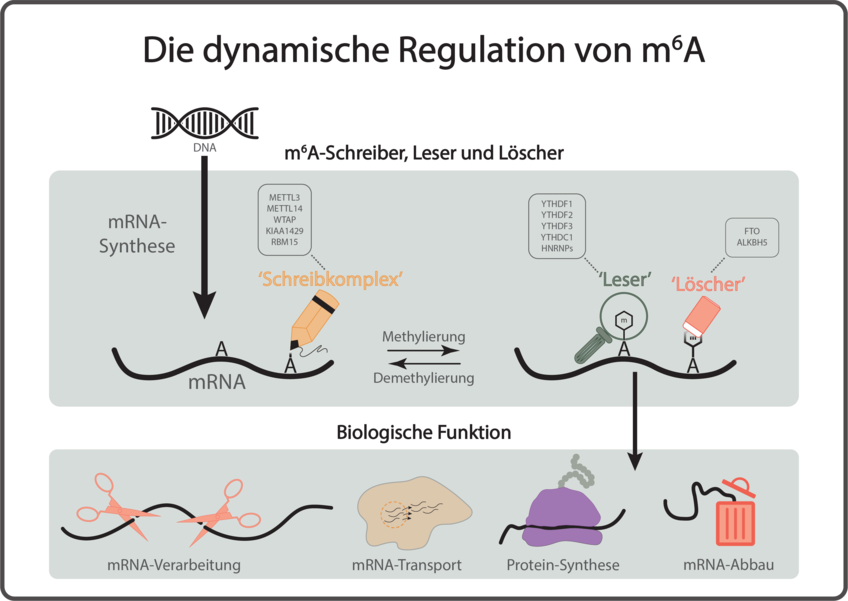
Abb. 1: Die mRNA-Methylierung m6A wird durch einen aus mehreren Untereinheiten bestehenden Schreibkomplex vorgenommen. Dieser Prozess ist reversibel und m6A kann durch spezifische “Löscher” wieder entfernt werden. Abhängig vom Methylierungs-Status einer mRNA können m6A-“Leser“ an die mRNA binden und unter anderem den Transport, die Verarbeitung und den Abbau der mRNA sowie die Effektivität der Proteinsynthese verändern.
Bisher sind über 100 verschiedene RNA-Modifikationen beschrieben [2]. Da ihre Untersuchung technisch anspruchsvoll ist, konzentriert sich die Forschung bisher auf einige wenige. Die am besten charakterisierte mRNA-Modifikation ist N6-Methyladenosine (m6A). Sie entsteht durch die enzymatische Methylierung eines Adenosins. Die Menge an Adenosin, die auf diese Weise modifiziert ist, wird dynamisch durch einen spezifischen m6A-Schreibkomplex (m6A-Methyltransferasekomplex) und zugeordnete m6A- “Löscher“ (m6A-Demethylasen) reguliert [3]. Letztere agieren wie molekulare Radierer und entfernen die Methylierung rückstandslos vom Adenosin (Abb.1). Der Methylierungsstatus einer mRNA stellt eine zusätzliche Informationsebene dar, die ihre weitere Verarbeitung beeinflusst. So kann m6A zum Beispiel zu einer Veränderung der dreidimensionalen mRNA-Struktur führen oder eine Bindestelle für m6A-spezifische Leseproteine bieten [3].
mRNA-Methylierung reguliert die Zellentwicklung und neuronale Signalübertragung
m6A findet sich in Hefen, Pflanzen, Insekten und Wirbeltieren [3]. Die Bedeutung von m6A für tierisches Leben wird dadurch unterstrichen, dass ohne m6A-Regulation embryonales Leben nicht möglich ist [2]. Zu den zellulären Prozessen, die bisher mit m6A in Verbindung gebracht wurden, gehören unter anderem die Erhaltung und Differenzierung embryonaler Stammzellen, die zelluläre Hitzeschockantwort und die Regulation neuronaler Aktivität [3; 4; 5].
Die Untersuchung von m6A im Mäusehirn hat gezeigt, dass seine Menge im Laufe der Gehirnentwicklung stark ansteigt und den höchsten Pegel im Erwachsenenalter erreicht [4]. Dabei unterscheiden sich die Menge und die m6A-Methylierungsmuster verschiedener Gehirnregionen [4; 5]. Die genauen Funktionen, die m6A im erwachsenen Gehirn erfüllt, sind Gegenstand aktueller Forschung. Bisher ist bekannt, dass m6A-regulierende Proteine Lernprozesse beeinflussen und die Kommunikation zwischen Nervenzellen durch Methylierung synaptischer mRNAs regulieren [4; 5].
Bei Patienten mit Depressionen ist die mRNA-Methylierungssignatur verändert
Eine vor Kurzem veröffentlichte Studie des Max-Planck-Instituts für Psychiatrie und des Max-Planck-Weizmann Labors für Stressforschung hat gezeigt, dass Stress die m6A-Signatur des Gehirns verändert [5]. Mäuse, die kurzfristig Stress ausgesetzt waren, wiesen schon nach wenigen Stunden eine Gehirnregionsabhängige Veränderung der m6A-Signatur auf. Um den zugrundeliegenden Mechanismus genauer untersuchen zu können, wurden genetisch manipulierte Mauslinien verwendet, die es ermöglichten, gezielt m6A-regulierende Proteine in erregenden Nervenzellen auszuschalten. Abhängig davon, welcher m6A-Regulator ausgeschaltet wurde, veränderte sich die Menge an m6A in den untersuchten Gehirnregionen. Mäuse, denen entweder ein m6A-Löscher (Fto), oder ein Teil des m6A-Schreibkomplexes (Mettl3) fehlte, zeigten Veränderungen in der Genexpression und in stressbedingten Verhaltensweisen: beide Mauslinien wiesen ein verstärktes Angstgedächtnis sowie Veränderungen im spontanen Grabverhalten auf.
Ob die Aktivierung des Stresssystems auch beim Menschen zu einer Veränderung in der m6A-Signatur führt, untersuchten die Wissenschaftler, indem sie gesunden Probanden und Patienten mit Depression das künstliche Stresshormon Dexamethason verabreichten. Der Vergleich der Blutproben zeigte, dass die Stressinduzierte m6A-Regulation bei Patienten mit Depression eingeschränkt ist. Dies deutet darauf hin, dass m6A eine Rolle bei stressbedingten psychiatrischen Erkrankungen spielen und eventuell als diagnostischer Biomarker verwendet werden könnte.
Die Erforschung epitranskriptomischer Markierungen verspricht Einsichten in Gehirnbiologie und Entstehung neuropsychiatrischer Erkrankungen
Die Erforschung der mRNA-Methylierung m6A zeigt, dass epitranskriptomische Markierungen eine bisher unerforschte Regulationsebene der Genexpression darstellen, die sowohl in physiologischen Prozessen als auch in der Pathophysiologie eine wichtige Rolle spielen. Zukünftige Studien werden die Funktion aller beteiligten m6A-Regulatoren sowie ihre Rolle bei stressbedingten psychiatrischen Erkrankungen genauer erforschen und darüber Aufschluss geben, ob m6A zur Diagnosestellung verwendet werden kann. m6A ist dabei nur eine von über 100 epitranskriptomischen Markierungen. Die Charakterisierung des gesamten Epitranskriptoms verspricht Einsichten in grundlegende Mechanismen der Gehirnbiologie und die Entstehung neuropsychiatrischer Erkrankungen.
Literaturhinweise
Cell 165 (3), 535-550 (2016)
Nucleic acids research 41 (D1), D262-D267 (2012)
Nature reviews Molecular cell biology 18, 31 (2017)
Journal of neurochemistry 147, 137-152 (2018)
Neuron 99(2), 389-403 (2018)